I Big Data e l’intelligenza artificiale stanno giocando un ruolo fondamentale per la lotta al coronavirus. Se da un lato i Big Data vengono utilizzati per monitorare “i furbetti della quarantena”, dall’altro potenti supercomputer lavorano ininterrottamente per cercare una possibile cura per il COVID-19.
Che cosa sono i dati anonimizzati?
I social network mettono a disposizione in forma anonima gli interessi, statistiche ed addirittura gli spostamenti dei propri utenti. Passando al setaccio questa mole di dati a disposizione è possibile, ricorrendo all’intelligenza artificiale, tracciare gli spostamenti delle persone negli ultimi giorni. Addirittura le maggiori compagnie telefoniche italiane, tra cui Tim, Vodafone, Wind Tre e Fastweb, hanno fornito un set di dati anonimi che aggregano gli spostamenti dei propri utenti.
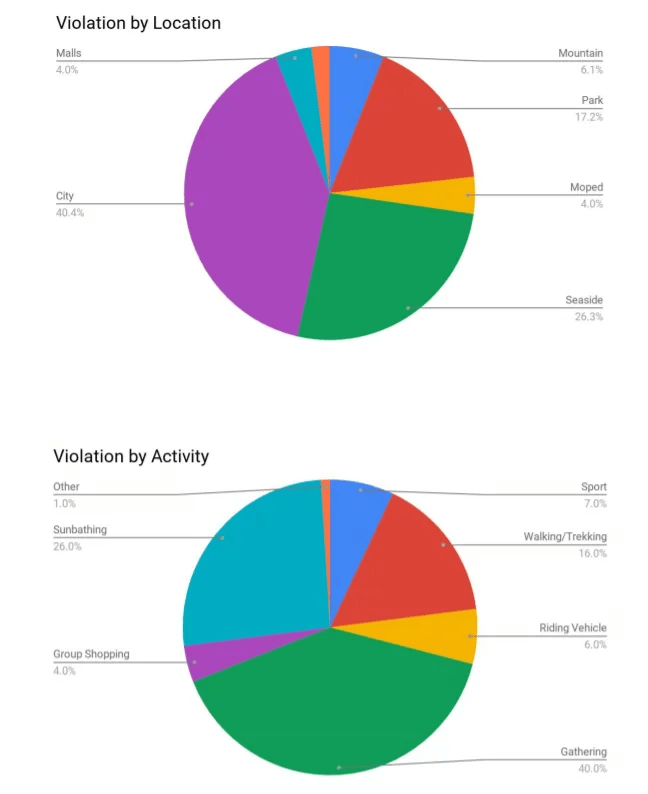
Dallo studio condotto sulle storie di Instagram tra l’11 e il 18 Marzo dai due gruppi di ricerca di Data analysis Ghost data e Logograb, è emerso che le regioni che hanno infranto le regole del decreto sono state la Campania e la Lombardia.
Lo studio ha rivelato anche cosa è stato fatto durante le violazioni: il 40% è andato in giro per la città, il 26% al mare, il 7% a fare sport, il 16% in montagna e il 4% nei supermercati in coppia.
Il supercomputer italiano per battere il coronavirus
I supercomputer di tutto il mondo stanno lavorando ininterrottamente per simulare la reazione del Sars-CoV2 alle molecole conosciute ed identificare quelle in grado di inibirne l’effetto.
E’ il caso del Cineca, il consorzio universitario con sede a Bologna, che grazie al progetto Exscalate4CoV, ha individuato 40 molecole che potrebbero giocare un ruolo fondamentale contro la lotta al Coronavirus.
Questo progetto è a trazione italiana e si è aggiudicato 3 milioni di euro del bando della Commissione Europea per progetti di ricerca sul Coronavirus nell’ambito del programma quadro Horizon 2020.
Il cuore di questo progetto è la piattaforma Exscalate che è stata sviluppata da una collaborazione tra Cineca, Dompè e Politecnico di Milano. Questa piattaforma ha una “biblioteca chimica” di 500 miliardi di molecole a cui i ricercatori possono attingere per simulare il comportamento del virus in abbinamento alle molecole farmaceutiche, ed è in grado di valutare di più di tre milioni di molecole al secondo, con costi e consumi energetici contenuti.
Stando a quanto riportato sul sito ufficiale del progetto, “Ogni proteina richiede almeno una settimana di simulazione continua su 16 nodi del supercomputer Cineca. Con un computer normale ci vorrebbero almeno 4 mesi per ogni proteina”.